-
-
-
- A01班
- A02班
システム癌新次元のソフトウェア
(このページは現在準備中です)
神戸に建設中の次世代スーパーコンピュータ(京速コンピュータ)「京」向けに開発されている「大規模遺伝子ネットワーク推定ソフトウェア SiGN (サイン)」等を利用して癌研究をすすめていきます.システム癌新次元ではヒトゲノム解析センターのスーパーコンピュータや京速コンピュータ「京」でこれらのソフトウェアを利活用し,がんの統合的理解を目指します.
大規模遺伝子ネットワーク推定ソフトウェア SiGN(サイン)
大規模遺伝子ネットワーク推定ソフトウェア SiGN(サイン)は DNA チップなどで得られる遺伝子発現データやタンパク発現データ,miRNA を含む non coding RNA 発現データなどから細胞内分子の発現制御システムのモデル化・予測を行うスーパーコンピュータ用ソフトウェア群です.SiGN は,時系列発現データからシステムをモデル化する状態空間モデルを用いた SiGN-SSM,時系列データおよびノックダウンや患者サンプルなどの静的データから遺伝子間の発現の依存関係をベイジアンネットワークを用いて予測する SiGN-BN,静的データから遺伝子間の発現の依存関係をL1正則化法を用いて予測する SiGN-L1 の 3 つの数学的・統計学的モデルを用いて大規模遺伝子ネットワークを予測・推定することができます.SiGN は非常に複雑で大量の計算を行うため,実行にはスーパーコンピュータが必須です.
SiGN-SSM
(準備中)
SiGN-BN
SiGN-BN はベイジアンネットワークを用いて遺伝子発現などの細胞内分子発現の依存関係を,観測されたデータから予測・推定するソフトウェアです.遺伝子ノックダウン実験や患者個人サンプルなどから得られる遺伝子・タンパク発現データから遺伝子間・タンパク間の発現の依存関係を表す遺伝子ネットワークを数学的な裏付けに基づき予測・推定します.薬剤を細胞に投与した際に得られる時系列発現データからは動的遺伝子ネットワークが予測可能で,観測時点毎のマスターレギュレータ遺伝子の活性を視覚的に捉えることも可能です.
ベイジアンネットワークはその計算量が膨大なことから,これまで数十〜数百遺伝子程度の小規模な遺伝子ネットワーク推定に限られて用いられて来ましたが,スーパーコンピュータの計算能力をフルに活用する新アルゴリズムにより数万遺伝子からなる遺伝子ネットワークの計算が可能になっています.また,観測データに対して最適なネットワーク構造を計算することは難しく,通常は局所最適解を求める発見的アルゴリズムが用いられてきましたが,スーパーコンピュータ上で動作可能な新アルゴリズムにより 30 遺伝子程度までは全体最適解を求めることも可能になっています.
このソフトウェアの入力データ
- DNA チップなどで得られる遺伝子発現データ
- 複数のサンプル・ノックダウン又は多時点の時系列データ
- 文献や他のネットワークなどから得られる遺伝子間の関係の事前情報(オプション)
このソフトウェアで得られる出力データ
- 遺伝子間の発現の依存関係を表す遺伝子ネットワーク
- 遺伝子,タンパク,miRNA などの分子を節点(ノード)とし,それを有効枝(エッジ)で結んだ有向グラフ
- 出力フォーマット
- Cell Illustrator / CanceriP で閲覧・解析・編集可能な CSML 形式
- タブ区切りプレーンテキスト
予測・推定可能な遺伝子ネットワークサイズ
- 全体最適解遺伝子ネットワーク:〜 31 遺伝子
- 局所解遺伝子ネットワーク:〜 1,000 遺伝子
- 全ゲノム遺伝子ネットワーク:〜 40,000 遺伝子
動的遺伝子ネットワーク可視化技術
SiGN-BN を用いることにより,時系列観測データから動的遺伝子ネットワークを予測・推定することが可能です.
この例はヒト肺腺癌細胞株 PC9 に上皮成長因子 EGF 及び抗がん剤 Gefitinib (イレッサ)を投与し,24 時間にわたって 1 時間毎に取得した時系列発現データ (観測点:0hr, 0.5hr, 1hr 以後 24hr まで 1hr 毎,全 26 時点.プラットフォーム:アジレント 4x44K DNA マイクロアレイ) から推定した動的ベイジアンネットワークによる動的遺伝子ネットワークです.Gefitinib 既知関連遺伝子 37 遺伝子(別色で左端に配置)を含む全 3,202 遺伝子からなり,各時点毎の変動遺伝子を抽出したものです.ノード(節点)の大きさはその遺伝子の各時点でのマスターレギュレータとしての影響度(hubness)の大きさを表します.動的遺伝子ネットワークの可視化によって重要遺伝子の時点毎の影響力の変化を確認できます.
SiGN-L1
(準備中)
パスウェイシミュレーション・遺伝子ネットワーク閲覧解析ソフトウェア Cell Illustrator Online (CIO)
上記のようなソフトウェアによって予測・推定されたネットワークを目で見えるように表示・解析するソフトウェアが Cell Illustrator です。
- ネットワークのレイアウト
- ネットワーク内の要素の色、形などの編集
- 異なる条件下から得られたネットワークの比較
- 注目する遺伝子の調査
などの機能をシステムがんで利用します。
Cell Illustrator はまた、遺伝子ネットワークを含めた生体内パスウェイ (シグナル伝達、代謝ネットワークなど) のシミュレーションができます。予測・推定されたネットワークの検証やそこから得られる仮説の提案などに利用します。
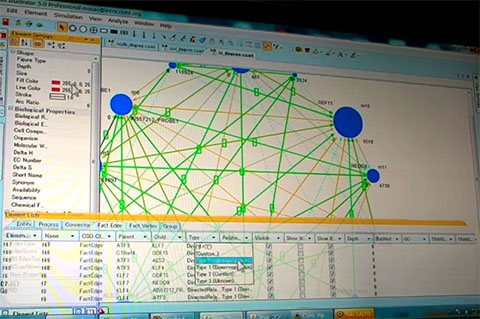 左図は推定した遺伝子ネットワークを表示している Cell Illustrator。 ある条件に適合する関係のコネクタを選択し、その色を編集しようとしているところ。
左図は推定した遺伝子ネットワークを表示している Cell Illustrator。 ある条件に適合する関係のコネクタを選択し、その色を編集しようとしているところ。
データ解析プラットフォーム Cancer integrative Pipeline (CanceriP)
上記の予測・推定ソフトウェアとその前後処理の作業手順をパイプラインフローとして組み立てることができるソフトウェアが CanceriP です。目的に応じたパラメータを設定済みのパイプラインフローを提供することで、一般のがん研究者にも遺伝子ネットワークの予測・推定が手軽にできるようになります。
また、Cell Illustrator では、遺伝子ネットワークを完全にインタラクティブな操作で解析・編集することができますが、解析・編集の対象の遺伝子ネットワークが多数になる場合などには面倒なことが想像できます。そのようなときに CanceriP で解析・編集の手順のパイプラインフローを組み立てることで、パイプラインフローによって決まりきった手順を繰り返し半自動的に行うことができるようになります。
 CanceriP では、 CSML の他に次のデータフローを作成できます。
CanceriP では、 CSML の他に次のデータフローを作成できます。
- 詳細なグラフ構造解析
- 統計解析システム R 言語での解析
- エクセルのようなデータ構造
それぞれのデータは相互に変換できます。
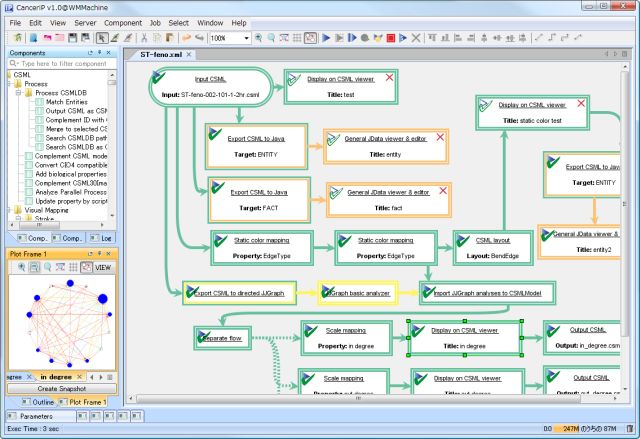
上図は推定した遺伝子ネットワークに対する処理のパイプラインフローを表示している CanceriP。ここでは、あるルールによってコネクタを色分けし、In degree, out degree, node degree などの簡単の分析の後、それらの情報をノードサイズに反映させる等の処理を行っている。